豊富な解析実績をもつ、メタゲノム株式会社の菌叢解析受託サービスです。
サービス概要
- 16S rRNA遺伝子解析、およびメタゲノム遺伝子解析の解析受託サービスです。
- 新鮮サンプル(糞便、スワブなど)、またはDNAサンプルから、NGSを用いた遺伝子配列解析を受託いたします。
- メタゲノム株式会社の豊富なノウハウと圧倒的な解析実績により最適化された、独自プロトコルおよび高精度解析パイプラインがご利用いただけます。
■常在菌叢のサンプリングからデータ解析までの高速・高精度解析パイプライン
■次世代シークエンサー(NGS)を用いた大量16S・メタゲノムデータ収集技術
■常在菌叢の正常さや異常さを数値化して評価する高度な情報学・統計学手法
■常在菌叢の全体構造の評価ならびに構成細菌の特定と存在量の定量解析
■常在菌叢の高品質データベースの構築技術
■個別微生物ゲノム解析技術
納品データについて
下記の内容をExcelにて納品させていただきます。
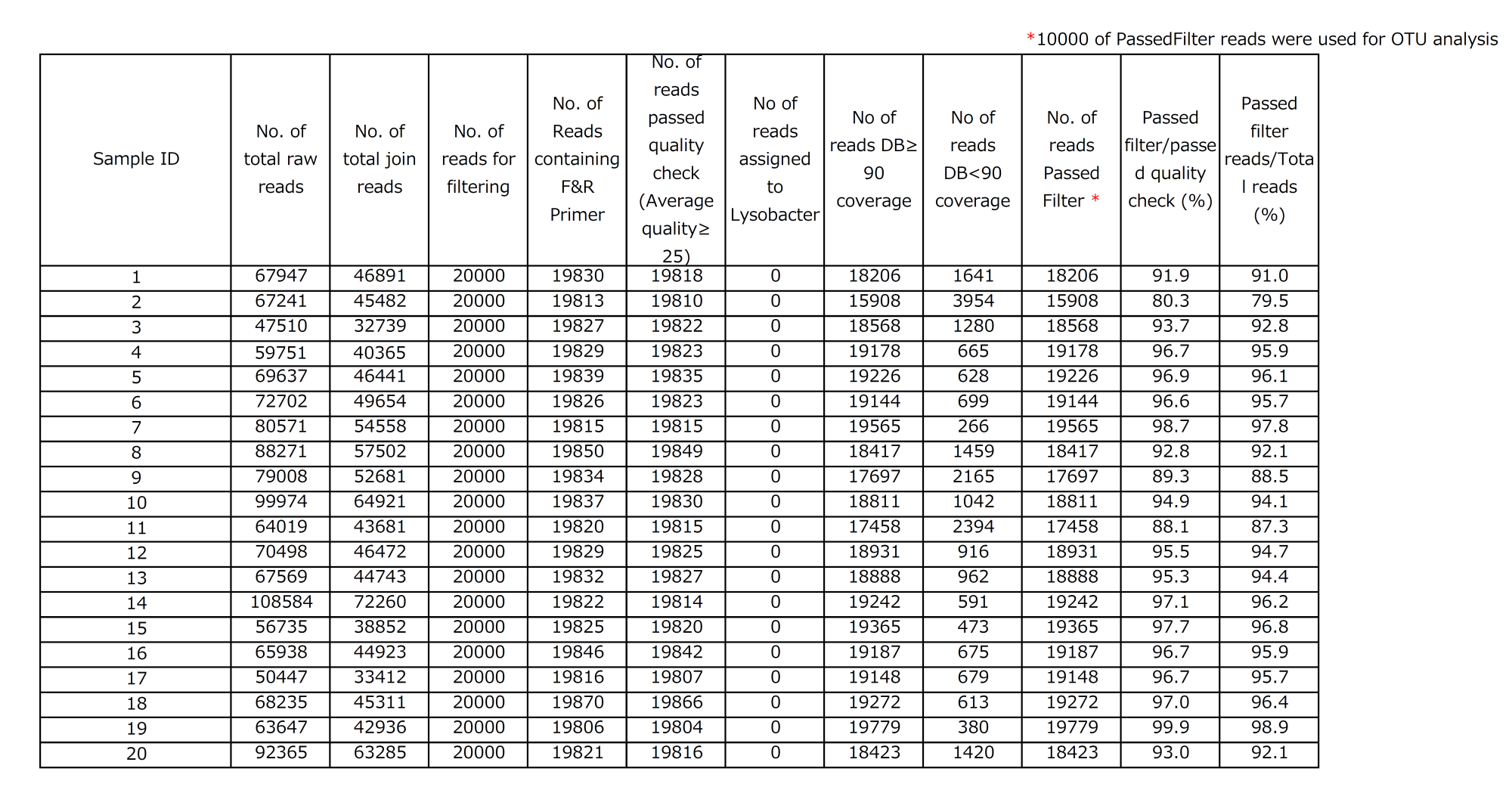
- Sample detail:サンプル識別コード
- Summary of Miseq reads:Miseqリードの要約(リード情報)
- α-diversity(α多様性):ひとつの環境における種多様性
- OTU assign:16S rRNA データベースに対する相同性検索による菌種の割り当て
- Phylum(門),Genus(属),Species(種)[ 菌種組成シート ]:菌種組成情報=サンプルごとの菌種の割合を表示します。
- UniFrac PCoA Weighted , UniFrac PCoA Unweighted (Unifrac距離) :OTU に含まれるリード数(組成比)を考慮した Weightedと考慮しない Unweighted UniFrac解析がある。
前者は菌種の有無と組成比の両方が両菌種間の類似性の距離に反映されますが、後者は菌種の有無のみとなります。
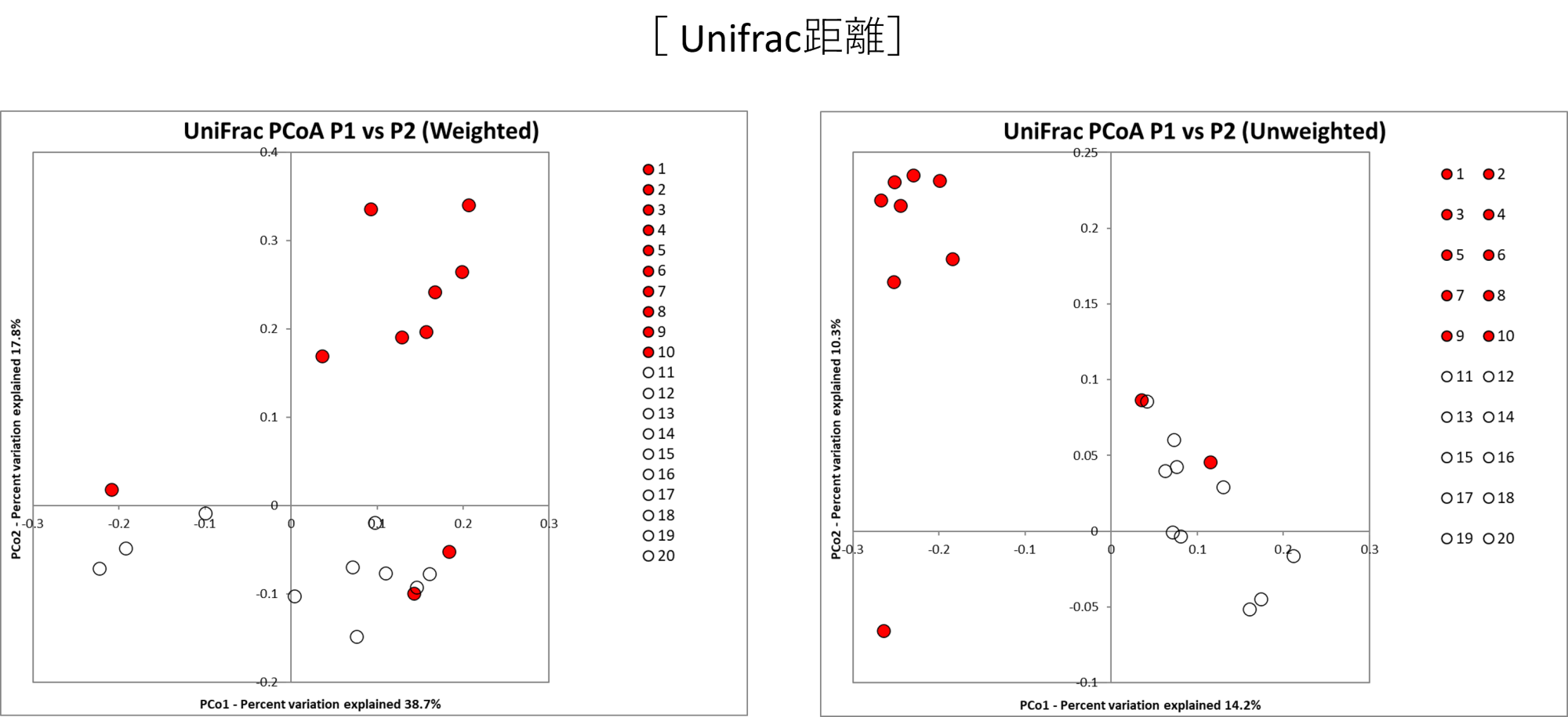
*上記納品データ例の表とUnifrac距離の図は、相関はございません。それぞれ結果の一例とご参照ください。
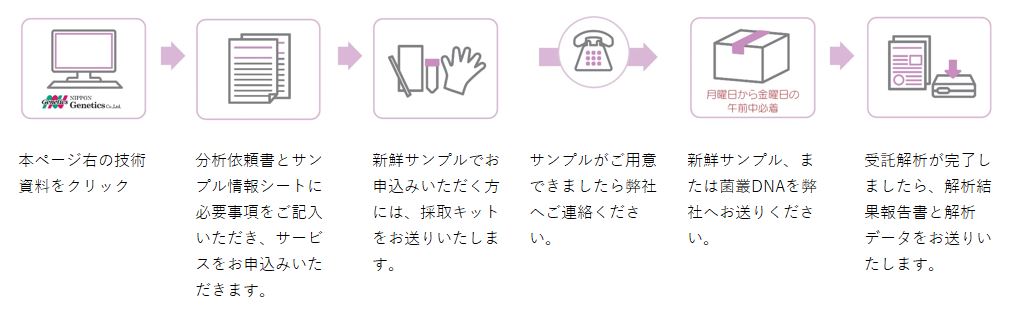
解析サービスの流れ
分析依頼書へ記入し、サンプルの送付をしていただくだけの簡単手続きで、ご利用いただけます。
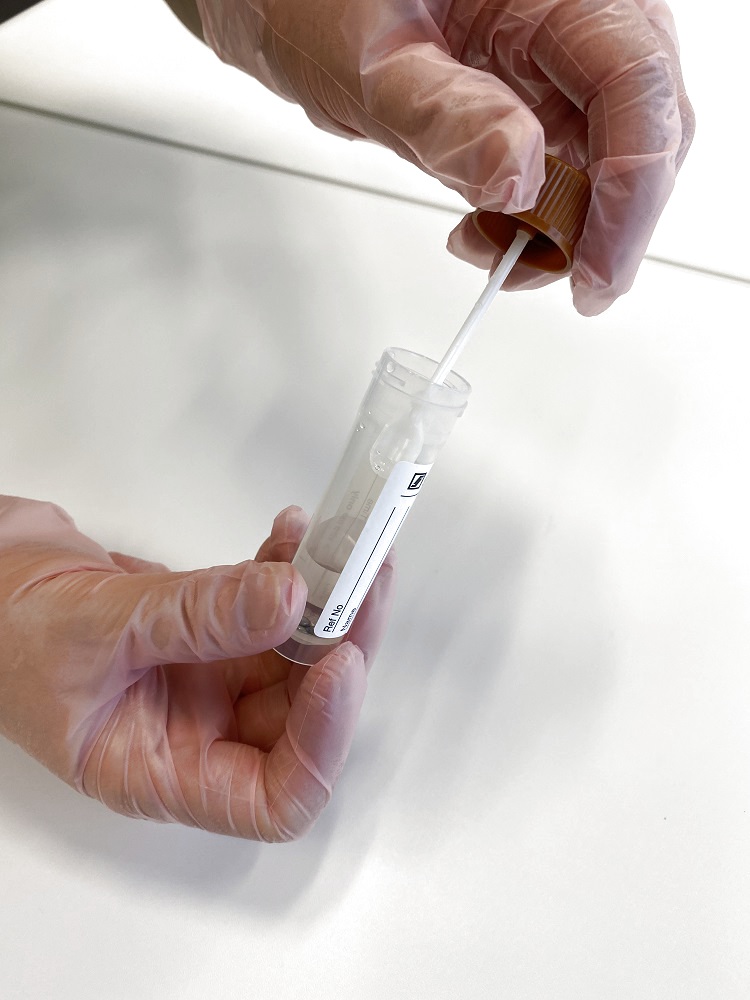
採取キット
糞便または唾液を採取して一時保管、輸送を行うための採取キットです。
※クリックで拡大します。
- チューブ内にRNA Later(RNA保存剤)が含まれているため一定期間(約1年)程度は、採取時の菌叢を維持したまま保管、輸送が可能です。
- 同様の効果がある液体窒素が不要です。
- 保管温度:4℃
- セット内容(1検体用/1本)
・採取容器
・RNA later 6 mL
・φ3 mmジルコニアビーズ
注意事項
- 納期:サンプル受付後2~3ヶ月となります。
- 受託可能数:10検体以上から承ります。
- 採取キット:10点以上のご注文から承ります。
- NGSはイルミナ社MiSeqを使用します。Ion PGM、IonProton、Sequelでの解析をご希望の場合はご相談ください。
- 受託可能な新鮮サンプルは、ヒトの糞便、唾液、皮膚、バイオプシ並びにマウス(動物)の糞便、バイオプシです。
- 植物、および環境細菌叢の新鮮サンプルは受託できませんが、DNAからの環境細菌叢のサンプルの分析は受託可能です。
- ヒト検体の解析をご依頼いただく場合は「人を対象とする生命科学・医学系研究に関する倫理指針」に基づいた
研究計画書、計画承認書、被験者への同意説明書などの書類をご提出いただく必要がございます。 - ヒト検体の解析をご依頼いただく場合は、新型コロナウイルス非感染証明書をご提出いただく必要がございます。
実績について
メタゲノム株式会社のデータ解析に関わる、共同研究先の研究実績
1.Aiyoshi T, Kakihara T, Watanabe E, Tanaka N, Ogata Y, Masuoka H, Kurokawa R, Fujishiro J, Masumoto K, Suda W.:
"A comprehensive microbial analysis of pediatric patients with acute appendicitis."
J. Microbiol. Immunol. Infect. S1684-1182(23)00074-9 (2023)
2.Kawamoto S, Uemura K, Hori N, Takayasu L, Konishi Y, Katoh K, Matsumoto T, Suzuki M, Sakai Y, Matsudaira T, Adachi T, Ohtani N, Standley DM, Suda W, Fukuda S, Hara E.:
"Bacterial induction of B cell senescence promotes age-related changes in the gut microbiota."
Nat. Cell Biol. 25(6):865-876 (2023)
3.Nagata N, Takeuchi T, Masuoka H, Aoki R, Ishikane M, Iwamoto N, Sugiyama M, Suda W, Nakanishi Y, Terada-Hirashima J, Kimura M, Nishijima T, Inooka H, Miyoshi-Akiyama T, Kojima Y, Shimokawa C, Hisaeda H, Zhang F, Yeoh YK, Ng SC, Uemura N, Itoi T, Mizokami M, Kawai T, Sugiyama H, Ohmagari N, Ohno H.:
"Human Gut Microbiota and Its Metabolites Impact Immune Responses in
COVID-19 and Its Complications."
Gastroenterology 164(2):272-288. (2023)
4.Furuhashi H, Takayasu L, Isshi K, Hara Y, Ono S, Kato M, Sumiyama K, Suda, W.:
"Effect of storage temperature and flash-freezing on salivary microbiota profiles based on 16S rRNA-targeted sequencing."
Eur. J. Oral Sci., 130(2), e12852 (2022)
5.Li Y, Watanabe E, Kawashima Y, Plichta DR, Wang Z, Ujike M, Ang QY, Wu R, Furuichi M, Takeshita K, Yoshida K, Nishiyama K, Kearney SM, Suda W, Hattori M, Sasajima S, Matsunaga T, Zhang X, Watanabe K, Fujishiro J, Norman JM, Olle B, Matsuyama S, Namkoong H, Uwamino Y, Ishii M, Fukunaga K, Hasegawa N, Ohara O, Xavier RJ, Atarashi K, Honda K.:
"Identification of trypsin-degrading commensals in the large intestine."
Nature 609(7927):582-589 (2022)
6.Kiguchi Y, Nishijima S, Kumar N, Hattori M, Suda W.:
"Long-read metagenomics of multiple displacement amplified DNA of low-biomass human gut phageomes by SACRA pre-processing chimeric reads."
DNA Res. 28(6):dsab019 (2021)
7.Masuoka H, Suda W, Tomitsuka E, Shindo C, Takayasu L, Horwood P, Greenhill AR, Hattori M, Umezaki M, Hirayama K.:
"The influences of low protein diet on the intestinal microbiota of mice."
Sci Rep. 10(1):17077 (2020)
8.Miyauchi E, Kim SW, Suda W, Kawasumi M, Onawa S, Taguchi-Atarashi N, Morita H, Taylor TD, Hattori M, Ohno H.:
"Gut microorganisms act together to exacerbate inflammation in spinal cords."
Nature 585(7823):102-106 (2020)
9.Takewaki D, Suda W, Sato W, Takayasu L, Kumar N, Kimura K, Kaga N, Mizuno T, Miyake S, Hattori M, Yamamura T.:
"Alterations of the gut ecological and functional microenvironment in different stages of multiple sclerosis."
Proc. Natl. Acad. Sci. U S A. 117(36):22402-22412 (2020)
10.Suzuki Y, Nishijima S, Furuta Y, Yoshimura J, Suda W, Oshima K, Hattori M, Morishita S.:
"Long-read metagenomic exploration of extrachromosomal mobile genetic elements in the human gut."
Microbiome 7(1):119 (2019)